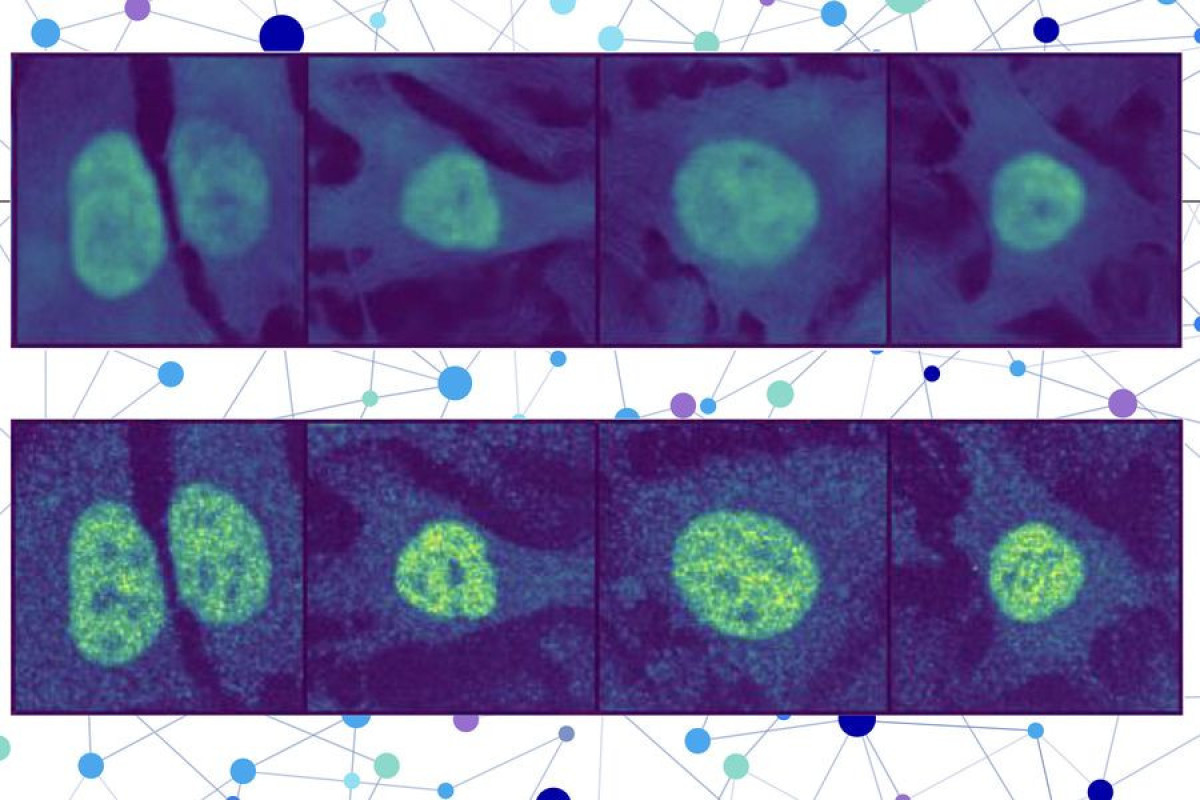
Επιστήμονες από το MIT, το Harvard και το Broad Institute ανέπτυξαν μια καινοτόμα μέθοδο μηχανικής μάθησης που επιτρέπει την ακριβή πρόβλεψη της θέσης μιας πρωτεΐνης μέσα σε οποιοδήποτε ανθρώπινο κύτταρο, ακόμα και σε επίπεδο μεμονωμένου κυττάρου. Η εξέλιξη αυτή αναμένεται να επιταχύνει τη διάγνωση ασθενειών, να ενισχύσει την ανακάλυψη φαρμάκων και να προσφέρει νέα γνώση στη βιολογία των πρωτεϊνών.
Η νέα μέθοδος, ονόματι PUPS, χρησιμοποιεί έναν συνδυασμό τεχνικών υπολογιστικής όρασης (computer vision) και γλωσσικών μοντέλων για να εντοπίσει πού βρίσκεται μια πρωτεΐνη μέσα στο κύτταρο, βασιζόμενη μόνο στη σειρά των αμινοξέων που τη σχηματίζει και σε τρεις μικροσκοπικές εικόνες από το κύτταρο. Το μοντέλο μπορεί να προβλέψει ακόμη και θέσεις πρωτεϊνών που δεν έχουν μελετηθεί ποτέ στο παρελθόν.
Ο ακριβής εντοπισμός των πρωτεϊνών είναι κρίσιμος για την κατανόηση πολλών ασθενειών. Η κακή τοποθέτηση μιας πρωτεΐνης στο κύτταρο μπορεί να προκαλέσει σοβαρές διαταραχές, όπως Αλτσχάιμερ, κυστική ίνωση και καρκίνο. Ωστόσο, με περίπου 70.000 διαφορετικές πρωτεΐνες ανά ανθρώπινο κύτταρο και περιορισμένες πειραματικές δυνατότητες, ο εντοπισμός τους είναι επίπονος και δαπανηρός.
Η υπάρχουσα βάση δεδομένων Human Protein Atlas έχει καταγράψει τη συμπεριφορά περίπου 13.000 πρωτεϊνών σε 40 κυτταρικές σειρές, καλύπτοντας μόλις το 0,25% των πιθανών συνδυασμών. Το PUPS φιλοδοξεί να καλύψει αυτό το κενό.
Η τεχνολογία βασίζεται σε δύο βασικά σκέλη:
1. Ένα γλωσσικό μοντέλο πρωτεϊνών, το οποίο αναλύει την αλληλουχία αμινοξέων και τη δομή της πρωτεΐνης.
2. Ένα μοντέλο υπολογιστικής όρασης τύπου inpainting, που επεξεργάζεται εικόνες από το κύτταρο (νουκλεόπλασμα, μικροσωληνίσκοι, ενδοπλασματικό δίκτυο) για να αναγνωρίσει το περιβάλλον του κυττάρου και να προβλέψει την πιθανή θέση της πρωτεΐνης.
Το σύστημα δημιουργεί τελικά μια εικόνα με επισήμανση του σημείου όπου βρίσκεται η πρωτεΐνη. Αυτό επιτρέπει τη μελέτη μεμονωμένων κυττάρων και όχι απλώς μέσων όρων ανά κυτταρική σειρά, κάτι που είναι καθοριστικό για εξατομικευμένες ιατρικές προσεγγίσεις, π.χ. στη στοχευμένη θεραπεία όγκων.
Σε αντίθεση με προηγούμενα μοντέλα, το PUPS δεν απαιτεί προηγούμενα δεδομένα για την πρωτεΐνη που αναλύει. Μπορεί να γενικεύει, δηλαδή να κάνει προβλέψεις για άγνωστες πρωτεΐνες και κυτταρικές σειρές. Μάλιστα, διακρίνει πώς διαφορετικά τμήματα μιας πρωτεΐνης επηρεάζουν τη θέση της, καθιστώντας το ένα χρήσιμο εργαλείο και για τη μελέτη μεταλλάξεων.
Κατά τη δοκιμή του μοντέλου, οι επιστήμονες προχώρησαν και σε πειραματική επιβεβαίωση: τα αποτελέσματα του PUPS συγκρίθηκαν με εργαστηριακά δεδομένα και παρουσίασαν μικρότερο ποσοστό σφάλματος από άλλες τεχνικές τεχνητής νοημοσύνης.
Ο συντονιστής της έρευνας, Dr. Yitong Tseo, τόνισε:
Μπορούμε να κάνουμε προβλέψεις για την τοποθέτηση πρωτεϊνών χωρίς να χρειάζεται να περάσουμε μήνες σε ένα εργαστήριο. Αυτό λειτουργεί ως αρχικός έλεγχος για το ποιες πρωτεΐνες αξίζει να μελετήσουμε περαιτέρω πειραματικά.
Οι ερευνητές σκοπεύουν να εξελίξουν το μοντέλο ώστε να μπορεί να προβλέπει αλληλεπιδράσεις μεταξύ πρωτεϊνών και να παρέχει προβλέψεις για πολλαπλές πρωτεΐνες ταυτόχρονα μέσα στο ίδιο κύτταρο. Μακροπρόθεσμα, το όραμα είναι να εφαρμοστεί το PUPS και σε ζωντανούς ανθρώπινους ιστούς, ανοίγοντας τον δρόμο για πρωτοποριακές εφαρμογές στην εξατομικευμένη ιατρική, την ογκολογία και τη φαρμακολογία.
[via]